检测到您当前使用浏览器版本过于老旧,会导致无法正常浏览网站;请您使用电脑里的其他浏览器如:360、QQ、搜狗浏览器的极速模式浏览,或者使用谷歌、火狐等浏览器。
 下载Firefox
下载Firefox
检测到您当前使用浏览器版本过于老旧,会导致无法正常浏览网站;请您使用电脑里的其他浏览器如:360、QQ、搜狗浏览器的极速模式浏览,或者使用谷歌、火狐等浏览器。
 下载Firefox
下载Firefox
2021年7月6日,北京大学生命科学学院生物信息中心(CBI)、北京大学生物医学前沿创新中心(BIOPIC)与北京未来基因诊断高精尖创新中心(ICG)高歌课题组,于Briefings in Bioinformatics上发表了题为“Identifying complex motifs in massive omics data with a variable-convolutional layer in deep neural network”论文,提出了基于自适应卷积核的新卷积学习模型vConv。
深度学习(Deep Learning)是机器学习的一种,通常指基于表示学习的深度神经网络,如基于卷积神经层构建的卷积神经网络、基于递归神经层构建的递归神经网络等。它适合用来发现海量高维数据背后的复杂模式。近十年来,随着计算机算力的大幅提升,深度学习在图像识别、自然语言处理等领域取得了众多成果,其中可以捕捉数据局部特征的卷积神经网络(Convolutional Neural Network, CNN)已被广泛应用于组学序列数据分析、生物影像处理等多个生命科学相关领域。
卷积神经网络可利用卷积层(Convolutional layer)中一系列卷积核(kernel)来自动识别输入序列上频繁出现的序列片段,并通过将这些片段组合以发现其中的序列motif。然而,目前卷积层只能使用预设固定长度的卷积核,难以适应海量组学数据中复杂多变的信号模式。为此,目前的主流实现中常用多种不同大小的卷积核分层叠加以应对,但由此导致的模型参数膨胀又显著提升了训练难度。
在本文中,研究人员提出了能够在训练中自动调整卷积核长度的新型变长卷积层vConv。vConv通过在原始卷积核上叠乘两条形状可训练的对向S型曲线,来动态遮蔽(mask)卷积核两侧元素、进而实时学习卷积核的有效长度(图一)。vConv可直接加入现有多层神经网络模型中,可作为传统卷积层的直接替代(drop-in replacement)广泛应用于数据挖掘、图像识别等多个领域。
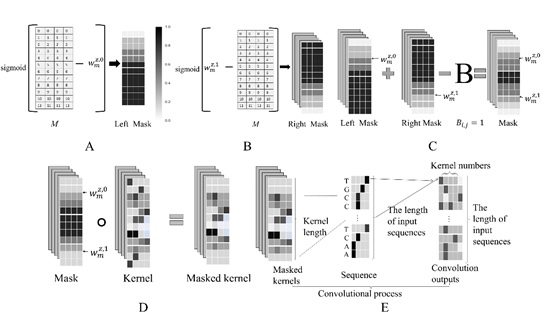
图一:vConv层结构。为了生成掩码矩阵(mask matrix),vConv使用两个对称的sigmoid函数生成了两个矩阵(A和B),然后通过叠加这两个矩阵获得了掩码矩阵(C)。在此基础上,vConv把该掩码矩阵与原始的卷积核做Hadamard积,获得了掩码内核(D),再将该掩码内核与输入序列进行卷积(E)。
序列motif(sequence motif)通常是指与特定生物学功能相关的一段序列片段、及其相关碱基/氨基酸分布模式,如转录因子结合位点、蛋白质功能域等。识别、鉴定与发现序列motif是生物信息学与计算生物学的经典问题之一,也是组学数据分析的重难点之一。研究人员发现基于vConv的神经网络可于Gb级别海量组学数据中准确识别鉴定序列motif,性能显著优于经典工具及基于传统卷积层的神经网络。
为方便使用,相关Python代码及教程已通过GitHub开源发布(https://github.com/gao-lab/vConv),与课题组前期发布的池化层ePooling方法(https://github.com/gao-lab/ePooling)相结合,可为相关应用提供了平滑的升级路径。
北京大学生命科学学院生物信息中心(CBI)、北京大学生物医学前沿创新中心(BIOPIC)与北京未来基因诊断高精尖创新中心(ICG)高歌研究员与课题组博士生丁阳(现为军事科学院军事医学研究院辐射医学研究所博士后)为共同通讯作者,北京大学生命科学学院博士生李静一、实习生金燊(现为美国卡内基梅隆大学计算生物学系硕士)为该论文的共同第一作者,北京大学生命科学学院本科生屠鑫明在代码测试上提供了大力支持。该研究得到了国家科技部、北京未来基因诊断高精尖创新中心、蛋白质与植物基因研究国家重点实验室的支持,计算分析工作于北京大学高性能计算校级公共平台与ICG高性能计算平台完成。