检测到您当前使用浏览器版本过于老旧,会导致无法正常浏览网站;请您使用电脑里的其他浏览器如:360、QQ、搜狗浏览器的极速模式浏览,或者使用谷歌、火狐等浏览器。
 下载Firefox
下载Firefox
检测到您当前使用浏览器版本过于老旧,会导致无法正常浏览网站;请您使用电脑里的其他浏览器如:360、QQ、搜狗浏览器的极速模式浏览,或者使用谷歌、火狐等浏览器。
 下载Firefox
下载Firefox
生命是细胞有序排列构成的整体。单个细胞在脱离生命体后难以独立发挥功能。因此需要联合细胞所处的微环境和空间位置来全面理解细胞功能。迅速发展的空间组学技术极大便利了在空间水平、单细胞精度上研究细胞功能以及组织中多细胞调控过程。特别的,对于不同空间组学切片之间的对齐和比较有助于揭示时间、疾病等变化过程中细胞在空间分布上的差异,解析背后的生物学机制。然而,目前的主流计算方法仍主要关注同质切片(如同一切片的空间域分割、同一组织多切片的重构等方面),难于处理跨时空、多模态的异构切片。
针对上述挑战,2023年11月9日,北京大学/昌平实验室高歌课题组于Nature Communications发表题为 Spatial-linked alignment tool (SLAT) for aligning heterogenous slices的研究论文,提出了跨平台、多模态空间组学比对与整合方法SLAT,可有效应用于跨组学空间对齐、连续时空发育解析等多种场景。

为有效处理异构切片,SLAT将空间数据建模为图,将细胞视为图中的节点、节点之间的边代表细胞之间的邻近关系,采用图神经网络聚合细胞的领域信息以获得基于空间的细胞表示,并通过对抗学习进一步消除图之间拓扑异质性,使得不同图中的细胞表示处在同一个空间中,从而实现跨平台、多组学异构切片之间的精准对齐。(图1)。
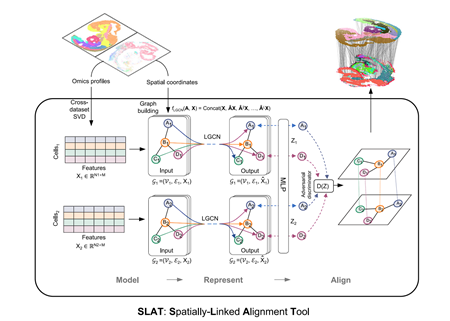
图1 SLAT模型的结构示意图
空间组学数据对齐的挑战之一在于不同空间组学技术平台的分辨率有着较大差异。例如,10x Visium的分辨率是50微米、Spatial ATAC-seq的分辨率是20微米、Stereo-seq的分辨率是0.2微米,差异达到了2个数量级。SLAT引入了可变临域概念,对于不同分辨率的技术在建图时采用不同的邻域大小,使得来自异质性空间数据的细胞的空间表示能够可比,从而在跨平台(Xenium与Visium,Stereo-seq与seqFISH)、跨组学(Stereo-seq与spatial-ATAC-seq)等异质数据的对齐上均有优秀的效果(图2)。
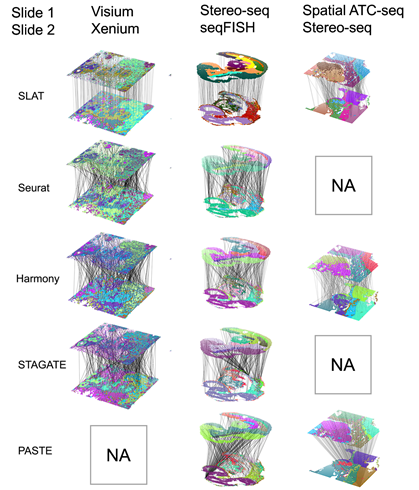
图2 SLAT在对齐跨平台,跨组学的异质数据优于其它方法
基于上述技术创新,SLAT可实现对如早期胚胎发育等复杂时空变化过程的精准追踪,如通过对小鼠发育E9.5-E16.5时期连续时空切片的解析(图3a),SLAT可以精准还原中肾(E11.5)和后肾(E11.5)发育为成熟肾脏(E12.5)的大尺度变化过程(图3b)。
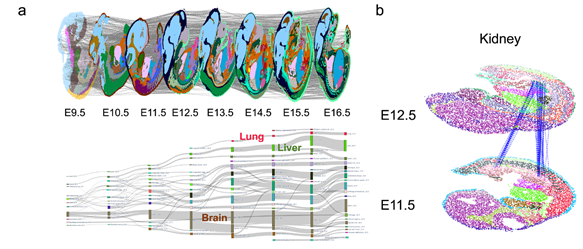
图3 SLAT可准确对齐不同发育阶段的时空切片数据
SLAT全部实现代码已经开源发布(https://github.com/gao-lab/SLAT),可通过PyPI平台直接安装使用。
北京大学生命科学学院博士生夏辰睿为该论文第一作者,生命科学学院本科生屠鑫明(现为华盛顿大学计算机科学与工程学院博士生)为论文的共同作者,“博雅博士后”曹智杰博士为该论文共同通讯暨共同第一作者。该研究得到了国家重点研发计划、蛋白质与植物基因研究国家重点实验室、北京未来基因诊断高精尖创新中心和昌平实验室的资助。计算分析工作于昌平实验室高性能计算平台、北京大学太平洋高性能计算平台与北京大学高性能计算校级公共平台完成。
论文链接:https://doi.org/10.1038/s41467-023-43105-5