检测到您当前使用浏览器版本过于老旧,会导致无法正常浏览网站;请您使用电脑里的其他浏览器如:360、QQ、搜狗浏览器的极速模式浏览,或者使用谷歌、火狐等浏览器。
 下载Firefox
下载Firefox
检测到您当前使用浏览器版本过于老旧,会导致无法正常浏览网站;请您使用电脑里的其他浏览器如:360、QQ、搜狗浏览器的极速模式浏览,或者使用谷歌、火狐等浏览器。
 下载Firefox
下载Firefox
真核细胞RNA聚合酶的转录活性于1959年由Samuel Weiss课题组利用大鼠肝脏核抽提物和四种rNTP在体外首次检测到1。 1969年,当时还在读研究生的美国生物化学家Robert Roeder 利用高盐溶液在海胆胚胎抽提物中发现、分离和纯化了三种形式的RNA聚合酶,并且按照它们在层析柱中洗脱出来的顺序依次命名为RNA聚合酶I、II、III2。1970年,Chambon课题组利用α-鹅膏蕈碱(a-amanitin)抑制实验证实存在三种不同活性的RNA聚合酶3。以蛋白质生成过程为例,核糖体RNA主要是由Pol I转录,核糖体蛋白的mRNA是由Pol II 转录,tRNA是由Pol III 转录。其中的一个关键,并且悬而未决的问题是:这三种RNA聚合酶如何协调以确保不同转录产物间的平衡。另外一个有意思的问题是Pol III主要转录tRNA, 但是其突变会导致不同组织的发育异常,也就是Pol III的转录如何产生组织特异性的影响?
2022年11月28日,北京大学季雄课题组在Genome Biology杂志在线发表了题为“Cross‑regulome profiling of RNA polymerases highlights the regulatory role of polymerase III on mRNA transcription by maintaining local chromatin architecture”的研究论文。研究者首次在同一种细胞内鉴定了三种RNA聚合酶的结合位点,研究了三者在全基因组水平上的交叉调控关系。研究者发现Pol III 对Pol II 转录调控影响最为广泛,鉴于对现有交叉调控机理的系统梳理发现至少有50%以上的基因表达变化是先前模型不能解释的,研究者提出并证实了Pol III-FACT通过局部染色质结构调控Pol II转录速率的新型机制。
研究者首先在小鼠胚胎干细胞中通过染色质免疫沉淀实验(ChIP-seq)对Pol I, Pol II, Pol III的染色质定位进行了系统鉴定,发现三者除了分别在各自转录基因区存在特异性结合外,还在基因组上存在数百个临近结合位点。进一步通过Pol I, Pol II, Pol III的诱导性蛋白质快速降解系统(degron),研究者分别降解其中一个进行另外两个的ChIP-seq实验,发现Pol III降解对Pol II的染色质结合的影响是其中变化最大的一组(图1)。而且Pol I, Pol II, Pol III降解前后的新生RNA检测实验(PRO-seq),也证实Pol III降解会造成最多Pol II peak区的转录变化,因此研究者首要研究了Pol III对Pol II的转录调控作用。
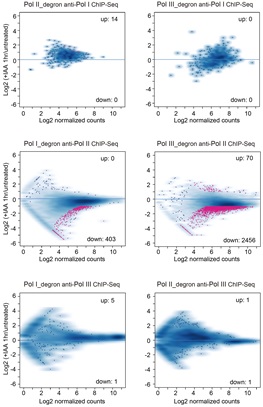
图1. 交叉ChIP-seq实验发现Pol III可以大规模影响Pol II基因转录
研究者通过分析Pol III 降解的PRO-seq和Pol II ChIP-seq信号在基因不同区域的变化情况,发现Pol III降解造成Pol II在一类基因内部区域的结合和转录信号增加,并且Pol III的其它亚基的降解也会造成了类似的Pol II转录变化,证实Pol III对Pol II介导的特异性基因的转录发挥调控作用。先前有三种模型来解释不同RNA聚合酶之间的交叉调控(图2):1)转录干扰模型认为不同RNA聚合酶转录的基因存在重叠时,干扰彼此间的转录过程;2)成环模型认为不同RNA聚合酶转录的基因间可以通过形成染色质环彼此促进转录;3)非编码RNA模型认为RNA聚合酶转录的非编码RNA可以直接调控另一种RNA聚合酶转录机器的组装。研究者发现至少有50%的Pol III 影响的Pol II转录基因不能通过上述模型解释。
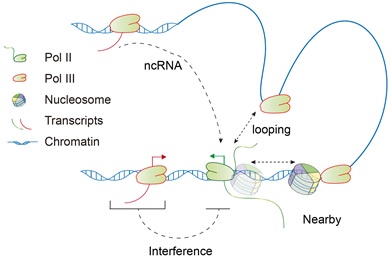
图2. Pol III 与Pol II 交叉调控的转录干扰(Interference), 成环(Looping)和非编码RNA (noncoding RNA)模型。
为了进一步探究这类调控背后的分子机制,研究者首先比较了Pol III在染色质上的结合和其发挥调控作用的这类基因的位置关系,发现Pol III在其调控的这类Pol II转录基因的启动子附近都有特异性结合,但是这些Pol III结合的位点并不是tRNA,因此意味着这些非编码区结合的Pol III发挥着对Pol II的调控作用。通过检测Pol III降解造成的染色质开放性和核小体占位变化(ATAC-seq),研究者发现Pol III降解特异性的造成调控基因的开放性降低,核小体占位增加。因此Pol III通过在非编码区的结合,造成临近的Pol II基因区的核小体占位发生变化,从而调控基因转录。结合定量质谱实验,研究者鉴定出了Pol III降解造成FACT复合体与Pol II的互作减少,从而造成Pol II转录区的染色质结构改变并导致其转录速率下降,从而导致Pol II在基因内部区域堆积,并通过FACT的功能缺失实验进一步确认了其在这一过程中发挥直接作用。
综上,这项工作系统刻画了不同RNA聚合酶的交叉调控关系,特别是Pol III通过在非基因区的结合,组织局部染色质结构,帮助组蛋白分子伴侣FACT复合体结合染色质,从而调控Pol II在多种功能基因区的转录速率(图3)。本研究也为认识RNA聚合酶的转录交叉调控乃至RNA聚合酶III功能失常带来的多种组织发育异常提供了新的视角。
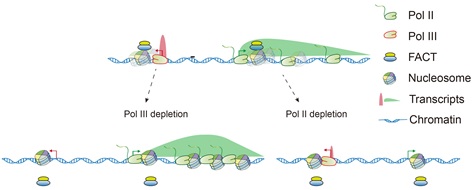
图3. Pol III 通过FACT和局部染色质结构调控临近Pol II基因的转录速率模型
北京大学生命科学学院,北大-清华生命科学联合中心季雄研究员是本文的通讯作者。北京大学生命科学学院蒋永鹏博士、黄捷博士为本论文共同第一作者。西湖大学郭天南课题组为研究提供了重要帮助。该工作得到科技部国家重点研发计划、国家自然科学基金、启东创新基金、北大-清华生命科学联合中心和细胞增殖与分化教育部重点实验室和北京大学博雅博士后基金的资助。感谢北京大学凤凰工程多个仪器平台对本项目的大力支持。
季雄课题组长期从事RNA聚合酶亚基功能调控和疾病机理的研究。主要集中在RNA聚合酶相关的新型功能、脑部疾病和核酸适配体筛选等方向,近期成果发表在Molecular Cell、 Nature Communications、Genome Biology(2)、Cell Discovery、CMLS、iScience和Method等杂志上,为选择性基因表达调控提供新的假说。现因发展需要,诚邀博士后和研究生加入。
原文链接: https://genomebiology.biomedcentral.com/articles/10.1186/s13059-022-02812-w
参考文献:
1. Weiss, S. B. & Gladstone, L. A mammalian system for the incorporation of cytidine triphosphate into ribonucleic acid1. Journal of the American Chemical Society, 81, 4118-4119 (1959).
2. Roeder, R. G. & Rutter, W. J. Multiple forms of DNA-dependent RNA polymerase in eukaryotic organisms. Nature 224, 234–237 (1969).
3. Kedinger, C., Gniazdowski, M., Mandel, J. L., Jr., Gissinger, F. & Chambon, P. Alpha-amanitin: a specific inhibitor of one of two DNA-pendent RNA polymerase activities from calf thymus. Biochemical and biophysical research communications, 38, 165-171 (1970).