检测到您当前使用浏览器版本过于老旧,会导致无法正常浏览网站;请您使用电脑里的其他浏览器如:360、QQ、搜狗浏览器的极速模式浏览,或者使用谷歌、火狐等浏览器。
 下载Firefox
下载Firefox
检测到您当前使用浏览器版本过于老旧,会导致无法正常浏览网站;请您使用电脑里的其他浏览器如:360、QQ、搜狗浏览器的极速模式浏览,或者使用谷歌、火狐等浏览器。
 下载Firefox
下载Firefox
近日,2021年度北京市科学技术奖揭晓,北京大学生命科学学院、蛋白质与植物基因研究国家重点实验室成员陆剑课题组与中国科学院昆明动物研究所吕雪梅课题组、中山大学吴仲义课题组、中国科学院上海巴斯德研究所崔杰课题组和中国医学科学院病原生物学研究所钱朝晖课题组共同完成的“新冠病毒谱系划分及进化动态分析体系的建立及应用”项目获2021年度北京市科学技术进步二等奖。
陆剑团队在疫情暴发早期就密切关注疫情进展,与崔杰等合作者于2020年初通过对当时公共数据库中仅有的103条新冠病毒基因组序列进行分子进化分析,发现参考基因组8782(orf1ab:U8517C,同义突变)和28144(ORF8:C251U,S84L)两个位点显示出显著的连锁。根据这两个高度连锁的位点,将SARS-CoV-2病毒分为L和S两个主要谱系(图1A)。以动物中发现的冠状病毒为外群,推测S谱系是祖先型,L由S谱系进化而来(图1B)。研究结果于2020年3月在National Science Review发表后,引起了广泛关注,被F1000推荐,该论文迄今被下载70余万次(编辑部提供数据),获《国家科学评论》2021年度优秀论文奖,入选中国科协第七届“优秀科技论文遴选计划”。S/L谱系划分也陆续被国内外其他独立研究印证,为新冠病毒基因组的谱系划分和演化分析提供了重要支撑。S/L划分系统被广泛应用于流行病学研究,被中国疾控中心等多家机构广泛用于疫情早期输入型病例的分子溯源,也被《中国—世界卫生组织新冠病毒溯源联合研究》中英文报告所采用。
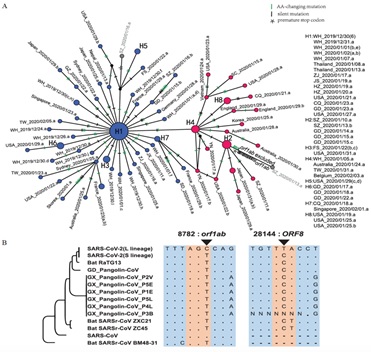
图1 新冠病毒的单倍型分析及进化关系
在S/L谱系划分基础上,陆剑团队与吕雪梅等课题组合作,进一步构建新冠病毒分层次谱系划分系统,揭示新冠病毒进化轨迹。研究团队分析了121,618个高质量的病毒基因组中的单核苷酸变异(SNV),根据3个主导性SNV将L谱系划分为L1和L2两个主要亚谱系,再根据位点的突变频率和连锁性筛选出201个代表性SNV,进一步逐级划分出130个亚谱系,绘制了反映各个谱系之间亲缘关系的单倍型网络图(图2),并建立了实时更新的配套网站(www.covid19evolution.net)方便查看亚谱系信息。该研究囊括了目前出现的绝大多数新冠基因组变异并整理了它们的谱系关系,阐明其中各个谱系时空分布的规律,搭建出新冠病毒演化的大体框架,为理解病原体变异规律和预测病毒的演化方向提供参考。该工作于2021年在Science Bulletin发表后,获该期刊年度“十大下载论文奖”(“Science Bulletin2021 Top 10 Downloaded Paper Award”)。
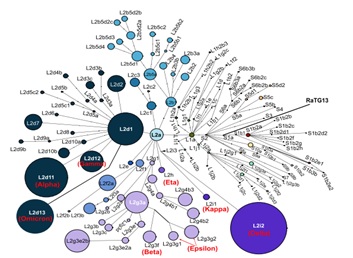
图2 新冠病毒亚谱系的单倍型网络
在病毒传播过程中,少数病毒颗粒散布就能引发新的疫情,这称为奠基者效应。以往病毒的奠基者效应大多建立在生态学模型或者病毒学模型上,很少考虑到病毒变异在这个过程中的作用。L和S谱系划分为研究奠基者效应提供了新的维度。项目组的陆剑、吴仲义和吕雪梅团队根据S和L谱系在不同国家的分布存在的差异,将经典的群体遗传学理论拓展至新冠病毒进化研究,首次将分支过程(branching process)应用到新冠病毒的传播研究中(图3)。通过模拟及真实数据拟合,提出5–10个带病毒的入境者足以引起大部分国家/地区的疫情这一观点。该研究发表在National Science Review(Ruanet al. 2021),为新冠病毒的科学防控提供了理论基础。
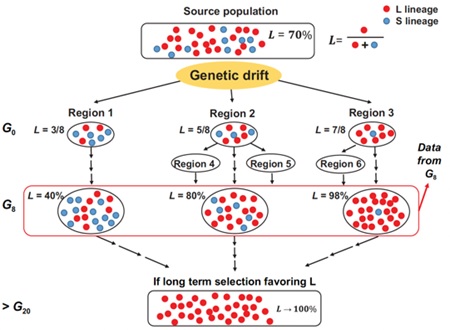
图3 新冠病毒传播的奠基者效应模型
为了进一步揭示新冠病毒流行株背后的进化推动力,陆剑团队与中山大学吴仲义团队合作,开展群体遗传模拟和大数据分析。模型分析表明,随着新冠疫情的不断扩散,病毒群体大小会随着感染人数的增加而变大,群体大小的扩大则会提高适应性变异出现和固定的概率,而加速的适应性进化速率反过来会加快病毒的扩散进而导致病毒群体的进一步增大,如此便会形成群体大小和进化速率之间的正反馈循环,最终导致病毒的失控演化(图4)。研究表明,随着新冠病毒感染人数的不断增加,新冠病毒于2020年底开始进入加速进化的阶段,导致高度进化的病毒株的快速涌现(Ruanet al., 2022,Molecular Biology and Evolution)。

图4 新冠病毒突变数目和感染人数随时间的变化动态
此外,陆剑团队与合作者围绕新冠病毒的起源和进化规律开展了一系列工作。与吕雪梅研究组合作,通过理论模拟,澄清了蝙蝠和穿山甲的病毒序列作为人类新冠病毒进化分析外群的可靠性方面的争议(Li et al. 2020,Science China Life Sciences)。与医学科学院病原生物研究所钱朝晖团队合作,撰写综述总结新冠病毒基因组分子演化动态和规律(Qian et al., 2022,Medical Review)。上述工作加深了对病毒基因组变异规律及演化规律的认识,对于疫苗和药物的研发也有重要的参考价值。
发表文章:
1. Tang X#, Wu C#, Li X#, Song Y#, Yao X, Wu X, Duan Y, Zhang H, Wang Y, Qian Z, Cui J*, Lu J* (2020) On the origin and continuing evolution of SARS-CoV-2. National Science Review 7(6): 1012–1023.
2. (2020) The use of SARS-CoV-2-related coronaviruses from bats and pangolins to polarize mutations in SARS-Cov-2. Science China Life Sciences 63: 1608–1611.
3. Tang X#, Ying R#, Yao X#, Li G, Wu C, Tang Y, Li Z, Kuang B, Wu F, Chi C, Du X, Qin Y, Gao S, Hu S, Ma J, Liu T, Pang X, Wang J, Zhao G, Tan W*, Zhang Y*, Lu X*, Lu J* (2021) Evolutionary analysis and lineage designation of SARS-CoV-2 genomes. Science Bulletin 66(22): 2297–2311.
4. Ruan Y, Luo Z, Tang X, Li G, Wen H, He X, Lu X*, Lu J*, Wu CI* (2021) On the founder effect in COVID-19 outbreaks: how many infected travelers may have started them all?. National Science Review 8(1): nwaa246.
5. Ruan Y#, Hou M#, Tang X#, He X, Lu X, Lu J*, Wu CI*, Wen H* (2022) The runaway evolution of SARS-CoV-2 leading to the highly evolved Delta strain. Molecular Biology and Evolution 39(3): msac046.
6. Qian Z*, Li P, Tang X, Lu J* (2022) Evolutionary dynamics of the severe acute respiratory syndrome coronavirus 2 genomes. Medical Review 2(1): 3–22.