检测到您当前使用浏览器版本过于老旧,会导致无法正常浏览网站;请您使用电脑里的其他浏览器如:360、QQ、搜狗浏览器的极速模式浏览,或者使用谷歌、火狐等浏览器。
 下载Firefox
下载Firefox
检测到您当前使用浏览器版本过于老旧,会导致无法正常浏览网站;请您使用电脑里的其他浏览器如:360、QQ、搜狗浏览器的极速模式浏览,或者使用谷歌、火狐等浏览器。
 下载Firefox
下载Firefox
2021年6月8日,北大-清华生命科学联合中心、北京大学生命科学学院伊成器课题组在Nature Methods杂志发表了题为“Detect-seq reveals out-of-protospacer editing and target-strand editing by cytosine base editors”的封面文章。在本研究工作的主要贡献如下:
1.建立了一种无偏向性的脱靶检测技术
作者通过捕获胞嘧啶碱基编辑器CBE ADDIN EN.CITE ADDIN EN.CITE.DATA [1]产生的中间体(dU),并进行一系列标记和富集 ADDIN EN.CITE ADDIN EN.CITE.DATA [2, 3],获得具有连续C-to-T突变的脱靶信号,并将其命名为Detect-seq(dU-detection enabled by C to T transition during sequencing)技术,该技术的主要技术路线如图1所示。

图- 1 Detect-seq 技术路线图
2.通过Detect-seq技术发现CBE工具导致的全基因组上的大量脱靶位点
研究人员在人胚肾细胞HEK293T和人乳腺癌细胞MCF-7转染BE4max ADDIN EN.CITE ADDIN EN.CITE.DATA [4],使用多种不同的sgRNA对基因组靶向位点进行C-to-T的编辑;同时对这些样本中进行了Detect-seq检测。通过Detect-seq检测发现,除RNF2位点外,其它靶向位点均可以产生几十至数百个Cas9依赖型的脱靶位点(图2)。
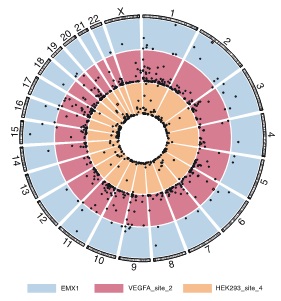
图-2 在三种不同sgRNA样品中鉴定到的Cas依赖型的脱靶位点 其中蓝色圆圈代表脱靶位点(off-targets),红色方块代表靶向位点(on-target) EMX1 n=48,VEGFA_site_2 n=511, HEK293_site_4 n=245
3.发现了两种新型脱靶位点(out-of-protospacer edit和target-strand edit)
有趣的是,研究人员基于Detect-seq技术还发现了两种新型的Cas依赖型的脱靶编辑:sgRNA结合区域外编辑(out-of-protospacer edit)及靶向链编辑(target-strand edit)(图3)。sgRNA结合区域外编辑即C-to-T的编辑发生在sgRNA结合区域外;而靶向链编辑则是C-to-T编辑发生在sgRNA与DNA结合链。根据Detect-seq的结果,上述两种编辑普遍存在于Cas9依赖型的脱靶位点附近。
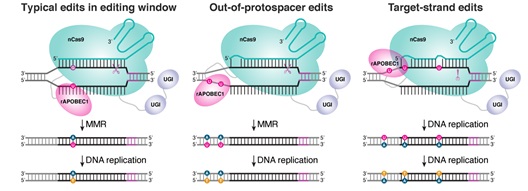
图- 3 sgRNA结合区域外编辑与靶向链编辑模式图
综上所述,Detect-seq技术为碱基编辑领域内提供了一种能够高灵敏、高特异、无偏好性地检测细胞内的CBE脱靶位点的方法。脱靶效应理解的加深,也将为阐明CBE的催化机制、优化改造更安全的CBE工具带来新的视角与可能。
北大-清华生命科学联合中心、北京大学生命科学学院伊成器教授为本文通讯作者;前沿交叉学科研究院雷芷芯博士,生命科学学院博士研究生孟浩巍、吕志聪为本文的共同第一作者;该研究得到了国家重点研发计划、国家自然科学基金以及生命科学联合中心的资助。凤凰工程北大基地和生命科学学院高性能计算平台为该研究提供了支撑。
原文链接:https://www.nature.com/articles/s41592-021-01172-w
博士后招聘:本实验室依托北大-清华生命科学联合中心和北京大学生命科学学院,通过化学生物学、单细胞组学、基因编辑等手段,对DNA和RNA修饰的组学技术、生物功能及其调控机制进行研究,尤其关注DNA/RNA生物学在人类疾病诊断与治疗中的应用。现诚聘表观遗传学、基因编辑及生物信息学博士后2-3名。详细信息请见链接:
http://www.bio.pku.edu.cn/homes/Index/news_cont/37/15652.html
1.Komor, A.C., et al., Programmable editing of a target base in genomic DNA without double-stranded DNA cleavage. Nature, 2016. 533(7603): p. 420-4.
2.Xia, B., et al., Bisulfite-free, base-resolution analysis of 5-formylcytosine at the genome scale. Nat Methods, 2015. 12(11): p. 1047-50.
3.Zhu, C., et al., Single-Cell 5-Formylcytosine Landscapes of Mammalian Early Embryos and ESCs at Single-Base Resolution. Cell Stem Cell, 2017. 20(5): p. 720-731 e5.
4.Thuronyi, B.W., et al., Continuous evolution of base editors with expanded target compatibility and improved activity. Nat Biotechnol, 2019. 37(9): p. 1070-1079.