检测到您当前使用浏览器版本过于老旧,会导致无法正常浏览网站;请您使用电脑里的其他浏览器如:360、QQ、搜狗浏览器的极速模式浏览,或者使用谷歌、火狐等浏览器。
 下载Firefox
下载Firefox
检测到您当前使用浏览器版本过于老旧,会导致无法正常浏览网站;请您使用电脑里的其他浏览器如:360、QQ、搜狗浏览器的极速模式浏览,或者使用谷歌、火狐等浏览器。
 下载Firefox
下载Firefox
近日,北京大学生命科学学院生物信息中心(CBI)、北京大学生物医学前沿创新中心(BIOPIC)、与北京未来基因诊断高精尖创新中心(ICG)高歌课题组,基于异构多组学数据,建立了人类RNA转录本编码能力(coding ability)跨细胞定量模型。相关论文已于近日以题为Quantitative model suggests both intrinsic and contextual features contribute to the transcript coding ability determination in cells在线发表于Briefings in Bioinformatics杂志。
基因的转录在生物学中心法则中起到了承上启下的作用,细胞通过转录-翻译过程多水平调控以应对环境压力和疾病。特别的,近年来研究显示,在传统编码蛋白的mRNA以外,部分RNA转录本(transcript)可以以非编码RNA形式发挥其生物学功能。综合运用生物信息学与计算基因组学手段,对转录本编码能力(coding ability)进行量化建模分析,不仅有助于深入理解转录-翻译这一基本的分子生物学信息传递过程,也可为精准解码细胞调控图谱提供重要的数据与方法学基础。
研究团队收集了发表于公共数据库来自22个不同细胞类型的人类Ribo-seq/RNA-seq配对数据并进行系统挖掘分析,对数据中101,170条转录本的翻译状态进行了严格判定。其中,46%的转录本为编码,43%为非编码。值得注意的是,他们发现11%的转录本,在不同细胞中呈现不同的翻译状态,即在部分细胞中编码,而在另一部分细胞中非编码。研究团队将其命名为“环境依赖编码转录本”(context-dependent coding transcripts, CDCTs)。
在此基础上,作者应用数据驱动的特征选择算法,综合运用序列内生(intrinsic, cis-)和细胞环境(contextual, trans-)特征,建立了人类RNA转录本编码能力跨细胞定量模型RiboCalc,实现了对人类转录本在多种细胞环境下的编码能力(即Ribo-seq表达量Ribo-TPM)的高精度预测(r = 0.81)。进一步模型分析显示,转录本的序列和所在细胞环境都对编码能力的决定起到了重要作用。值得注意的是,自14年以来即有若干工作报导一些传统被注释的非编码RNA可以在特定条件下结合核糖体甚至产生肽段(如 ADDIN EN.CITE ADDIN EN.CITE.DATA [1]),RiboCalc模型分析显示,这些RNA转录本与不结合核糖体的RNA转录本相比编码能力分数显著高,进一步提示转录本的编码能力不应被简化为单纯的编码/非编码二分分类,而是一个依赖于环境的连续定量指标。
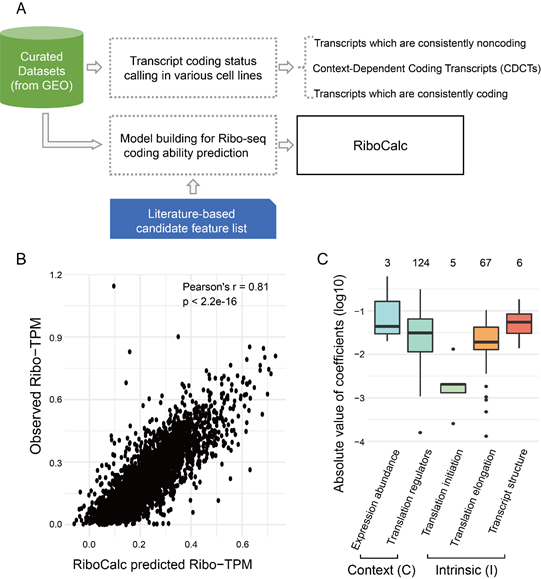
图一:RiboCalc工作流程与分析结果。A. RiboCalc模型构建流程。B. RiboCalc测试结果比较。C. RiboCalc各类特征的重要性。
作为首个针对高等哺乳动物开发的转录本编码能力定量预测模型,RiboCalc在人类中实现了跨组织、细胞类型的转录本编码能力量化建模与预测。目前,相关算法与教程已发布于https://github.com/gao-lab/RiboCalc/供相关领域海内外研究人员使用,相关问题可邮件联系ribocalc@mail.cbi.pku.edu.cn。
北京大学生命科学学院博士后亢雨笺为该论文的第一作者,高歌研究员为通讯作者,北京大学李静一、柯岚、降帅、杨德昌和侯玫在模型测试和数据分析上提供了大力支持。本研究工作得到了北京未来基因诊断高精尖中心ICG、蛋白质与植物基因研究国家重点实验室与国家重点研发计划“精准医学专项”的大力支持,计算分析工作于北京大学高性能计算校级公共平台和北京大学太平洋高性能计算平台完成。
参考文献:
1. Ruiz-Orera J, Messeguer X, Subirana JA et al. Long non-coding RNAs as a source of new peptides, Elife 2014;3:e03523.